SYBR qPCR Mix、Probe qPCR Mix, RT-PCR、PCR、 ...
more >>发布日期:2018-11-29 返回列表
由于qPCR实验操作简单,检测速度快,灵敏性高和特异性好等特点,qPCR已成为核酸定量实验的金标准。同时,其在其他领域中也有广泛应用,如病原菌、病毒微生物的定性定量检测,基因拷贝数检测,基因表达分析,基因融合分析,SNP分析,转基因食品中外源基因的鉴定,癌症复发的风险评估以及法医中的应用等,使其在生命科学,农学,分子诊断和医学中被广泛的应用。
前几期我们给大家分享了qPCR原理和发展历程、引物的设计流程以及实验方法的设计,这一期给大家分享的是核酸的提取及质控环节。样品的采集、处理和制备,核酸的提取及质控是开展qPCR实验的初始环节,也是决定qPCR实验结果准确与否的重要环节之一。正确的样品采集和处理方式,以及有效的核酸提取和严格的质控是qPCR实验结果准确的保障。那么根据MIQE,这里给大家一些指导和建议:
1.样本采集、处理和制备
样本的采集可能是实验变异的第一个来源,样本采取的量,部位以及时间是否统一是结果正确与否的重要考量,所以在样本采集的时候一定要强调量、部位和时间的一致性;另外采集的环境和保存方法也是非常关键的环节,特别是RNA实验,相对于DNA来说,RNA的易降解特性,因此容易被不当的样本采集方式和处理方式导致RNA的降解从而影响结果的准确性。对于RNA样品,建议样品采集时应在较洁净的环境下并低温或冰上操作,样品采集后应用液氮速冻并-80℃保存;如果用Trizol法提取RNA,可以将样品放置Trizol中,匀浆后-80℃保存;另外也可以将样品保存在RNA保护剂中(现在市面上有许多成品的kit),避免RNA的降解。那么对于DNA的样品来说处理方式和保存条件相对简单一些。
2.核酸提取
核酸的提取是第二个关键步骤,核酸的提取效率,提取的量以及纯度都会影响后续的实验结果。因此,选择正确的提取方法以及质控方法非常关键。
(1)DNA提取方法:DNA提取目前用的较多的方法有有机溶剂提取法 膜柱法 磁珠吸附提取法。
a.有机溶剂提取法 即酚/氯仿提取法。其主要利用DNA易溶于水而不溶于有机溶剂、蛋白质在有机溶剂存在时可变性沉淀的原理,根据核酸和蛋白对酚和氯仿变性作用的反应性不同分离核酸与蛋白后,在高盐的条件下利用乙醇沉淀收集DNA。该方法可对交大的组织样本进行提取,并获得相对较高的产量和质量。然而该方法比较费时费力,同时需要操作人员有一定的经验,无法大批量提取且难以自动化,另外,有机溶剂对操作人员的健康具有潜在危害。
b.膜柱法 该方法主要利用了DNA分子固相结合的原理,将DNA吸附于离心柱的吸附膜(如玻璃纤维素膜)上,同时离心除去蛋白质及RNA等分子。该方法可对多种样本类型进行提取,并获得优质的DNA(基因组及小片段DNA均可)以供后续的分析使用,由于膜柱法操作简便,适用于大规模和高通量处理。但是,当起始材料过的(如交大的组织样本)或不完全均质化时可导致吸附膜堵塞,从而导致产量降低或潜在的污染。
c.磁珠吸附提取法 生物磁珠即具有极小粒径的超顺微磁球,其具有丰富的表面活性基团,可以与各类生化物质偶联,并在外加磁场的作用下实现分离。根据磁珠上包被的基团不同,可将磁珠分为环氧基磁珠、氨基磁珠、羧基磁珠、醛基磁珠、巯基磁珠及硅基磁珠。其中,环氧基磁珠、氨基磁珠及羧基磁珠可用于各类蛋白或抗体的分离,巯基磁珠可用于重金属物质的分离。而用于DNA分离提取的磁珠则为硅基磁珠,其提取原理为利用氧化硅纳米微球的超顺磁性,在盐酸胍、异硫氰酸胍盐等和外加磁场作用下,DNA分子可被特异高效地吸附。该方法较膜柱法提取消除了样本堵塞膜的影响,同时操作简便易于自动化。此外,游离的磁珠与核酸的结合量更大,特异性的结合使得核酸纯度更高。
(2)RNA提取方法:目前RNA提取方法有Trizol提取法 膜柱法 磁珠吸附提取法。
a.Trizol提取法 该方法主要是利用了Trizol试剂含有苯酚、异硫氰酸胍等物质,能迅速破坏细胞并抑制细胞释放的核酸酶的特点,在异丙醇作用下可完整沉淀样本中RNA分子。该方法最为经典、传统,且适用于大多数样本类型,尤其是较难裂解的组织样本。但需注意的是该方法在操作过程中也有可能会引入影响后续PCR酶促反应的抑制剂(如血液中的血红蛋白,植物样本中的腐殖酸、黄腐酸等,以及在实验过程中带入的EDTA、肝素、氯酚、氯仿等)。这些以直接会对后续的反转录及qPCR等产生影响,导致结果的偏差。
b.膜柱法 该方法采用一系列裂解液裂解组织或细胞,并同时抑制RNA酶,硅胶模特异性吸附RNA后多次漂洗除去DNA、蛋白质及其他杂质,最后经低盐溶液洗脱RNA。与Trizol提取法相比,膜柱法更为简单且易于自动化,适用于大规模和高通量处理。
c.磁珠吸附法提取 根据使用磁珠类型的不同,该方法可分别对样本的总RNA及mRNA进行提取。磁珠法提取样本总RNA的原理与磁珠法提取样本DNA的原理基本相同,均为利用硅基磁珠对核酸的亲和吸附能力,在高盐环境和外磁场的作用下对核酸进行分离。但与DNA提取不同的是,在使用磁珠对RNA进行提取前,需采用特殊的裂解液对样本进行前处理以除去RNase并分离RNA层以进行后续总RNA提取。而与硅基磁珠提取又有所不同,磁珠法提取样本mRNA使用的是包被有亲和素的磁珠。将待提取的样本与生物素标记的oligo(dT)探针进行退火结合后在与包被有亲和素的磁珠相互作用即可达到分离mRNA的目的。采用磁珠法提取RNA较膜柱法消除了样本堵塞吸附膜的影响,同时操作更简便。但由于RNA吸附磁珠制备要求和成本较高,目前市场上商品化的试剂盒并不多。
3.核酸质量质控
a.为什么要做定量质控?
提取样本中的DNA/RNA浓度定量分析很重要,对于绝对定量,样品必须在标准曲线范围内。如图 1 所示,标准曲线为线性,但是这一趋势不是可以无限延伸的。一旦模板浓度超过 PCR 反应的限值,扩增效率将会降低,定量的线性下降。而模板浓度过低时,背景污染物可能被误认为是扩增信号,信噪比不足。同时,将标准曲线外推至更大范围也是具有风险的,所以,在进行qPCR 前最好确认样品在合适的浓度范围内。

对于相对定量,如通过 RT-qPCR 进行基因表达,较少的模板量会增大误差。如使用适当模板量进行分析,诸如SNP 基因分型等方法亦可生成更为可靠的定性数据。如图 2 所示,未知样品的信号强度应与标准样品类似。模板量过少将使等位基因表达量不可靠甚至无法使用。
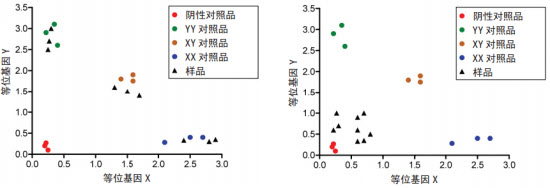
图 2:使用适当模板量(左图)和少模板量(右图)进行 SNP 基因分型的示例。当使用适当模板量时,未知样品的等位基因信号非常清晰,而模板量少时,等位基因信号会不可靠或无法使用。
另外,在比较不同样本时,使用相同数量的DNA/RNA是非常明智的。
常用的核酸定量质控方法
目前实验室最常用的几种量化方法,如分光光度法(quickdrop(MD),NanoDrop(Thermo)),微流控分析法(Agilent Technologies’ Bioanalyzer, Bio-RadLaboratories’ Experion),毛细管凝胶电泳法(Qiagen’s QIAxcel),或者荧光染料检测法。分光光度法原理为核酸、核苷酸及其衍生物都具有共轭双键系统,能吸收紫外光,RNA和DNA的 紫外吸收峰在260nm波长处。一般在260nm波长下,每1ml含1μg RNA溶液的光吸收值为0.022〜0.024,每1 ml含1μg DNA溶液的光吸收值约为0.020,故测定未知浓度RNA 或DNA溶液在260nm的光吸收值即可计算出其中核酸的含量。此法操作简便,迅速。若样品内混杂有大量的核苷酸或蛋白质等能吸收紫外光的物质,则测光误差较大,故应设法事先除去。NanoDrop及其它紫外分光光度计均采用紫外吸光度进行检测,它无法区分出 DNA、RNA、降解核酸、游离核苷酸及其它杂质。
微流控分析法(2100)通过微流体技术对样品进行分离。通过加入电压,使与荧光染料结合的样品在芯片上的显微蚀刻管道进行分离。根据核酸分子大小的不同而迁移率的不同进行分离,并通过激发光使染料发出荧光,使其能被仪器检测到。再根据已知的分子量及含量的Ladder计算DNA迁移时间和大小的函数关系,根据公式计算样品的分子量大小及浓度。
荧光染料检测法,它可以采用荧光染料检测特定目标分子的浓度。它采用专门研制的荧光检测技术,该技术采用只有与DNA、 RNA或蛋白质结合后才发出荧光的Molecular Probes ®染料。这些荧光染料只有与特异性的靶分子结合时,才能发出荧光信号,即使有游离核苷酸或降解核酸存在时亦是如此。由于染料法只检测靶分子(而不是污染物)的浓度,因此这种特异性可以获得十分精确的结果。
这些方法可以有不同的结果,因此用不同方法比较结果是不明智的。定量DNA/RNA推荐使用荧光结合染料法,该方法是检测低浓度样本最好的方法,同时也是几种定量方法中最准确的方法。建议任何情况下检测所有样本使用同一种方法。
b.纯度检测
提取过程中残留的污染物将大幅影响下游分析,而很多污染物可采用 quickdrop,NanoDrop 分光光度计检测出来。图 3 显示了纯化的 DNA 样品 (A),以及被胍 (B) 和苯酚 (C) 污染的同种样品。通过初步检测,两份污染样品均呈现核酸的特异性光谱特征,实际上苯酚污染样品的 260/280 比率多为正常。
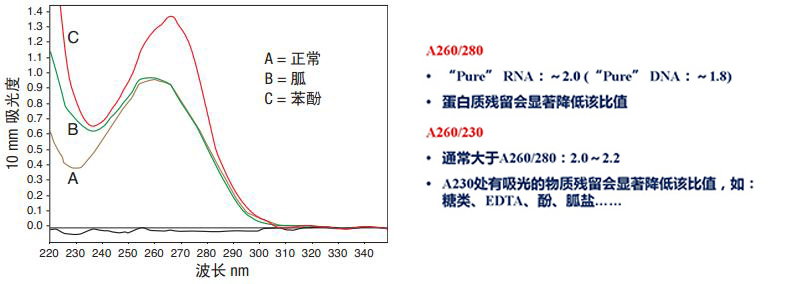
图 3 :无污染的纯化DNA ( A ),以及被胍( B )和苯酚( C )污染的同种DNA样品的光谱图。注意在有污染的样本中,其光谱图中波谷和波峰会发生变化,一般分别出现在230 nm 和 260 nm 处。
尽管大致光谱图无法诊断出问题,但是可能有助于鉴别问题是否存在并缩小问题来源的范围。许多污染物吸收发生在 230 nm 或更短的波长附近;有些污染物会给下游应用带来问题。除检测 260/280 比率和光谱的大致形状外,我们还推荐进行下列操作:
• 检查 260/230 比率 – 比率偏小表明样品中有污染物,而该污染物在 230 nm 或更短波长处存在吸收。
• 检查光谱中波谷处的波长 – 应该位于 230 nm 处。在短波处有吸收的污染物一般会使得波谷右移。
• 检查光谱中波峰处的波长 – DNA 和 RNA 应在 260nm处。在长波处有吸收的污染物一般会使得波峰位置右移。
有些污染物具有特征谱图,如苯酚。但是很多污染物的特征谱图相似:吸光度发生在 230 nm 或更短波长处。230 nm处的吸光度可能表明样品或提取过程出现问题,因此全面考虑这两方面十分重要。例如:较高的 A230 值(较小的A260/A230 比率)可能由如下原因导致:
• 多糖残留(通常出现在植物中);
• 核酸提取中残留苯酚;
• 残留胍(通常用于基于色谱柱的试剂盒);
• 用于核酸沉淀的糖原残留。
处理用分光光度计进行纯度检测外,还可以用电泳配合进行检测,可以通过电泳图看是否有RNA、DNA及蛋白污染。RNA样品中如果有基因组DNA污染,可以用DNase I进行处理,另外跨内含子进行引物设计也会减少基因组污染的影响(不知道您是否还记得我们第二期的内容呢);如果样品中含有蛋白等其他杂质的污染,推荐使用KAPA或贝克曼磁珠进行核酸纯化。
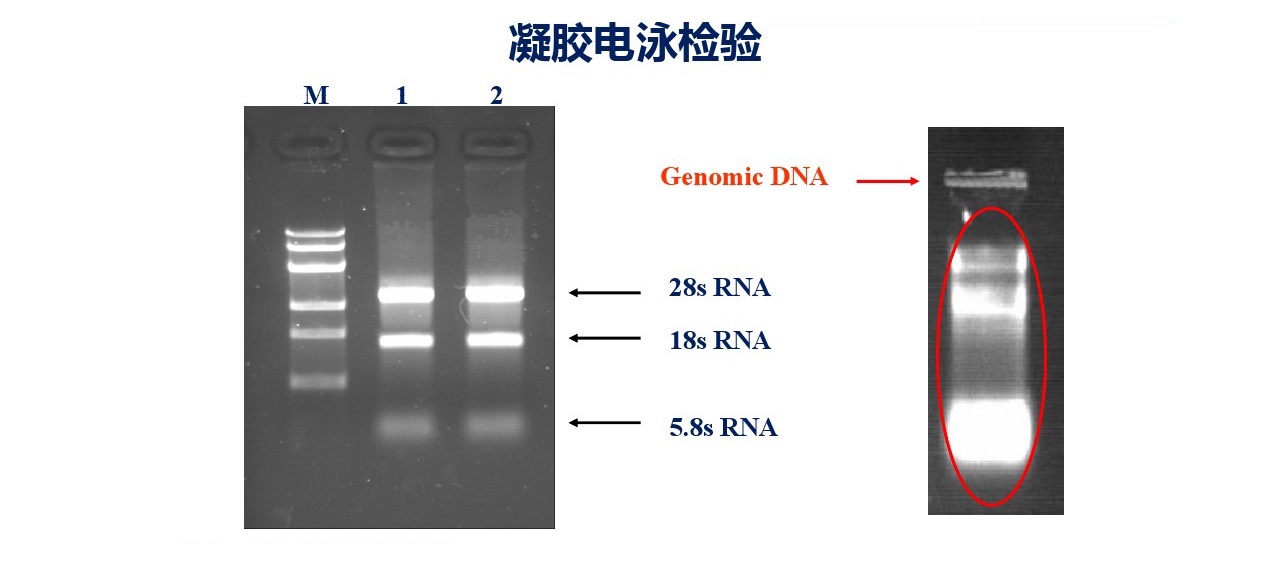
图4 :核酸凝胶电泳检测是否有RNA or DNA污染。
c.完整性检测
完整性检测是必不可少的一环,特比是对于RNA样品来说,RNA降解会大大影响我们的实验结果。对于完整性检测来说,我们可以用凝胶电泳检测,也可以用2100,LabChip进行检测,而后者更是完整性检测的金标准了。
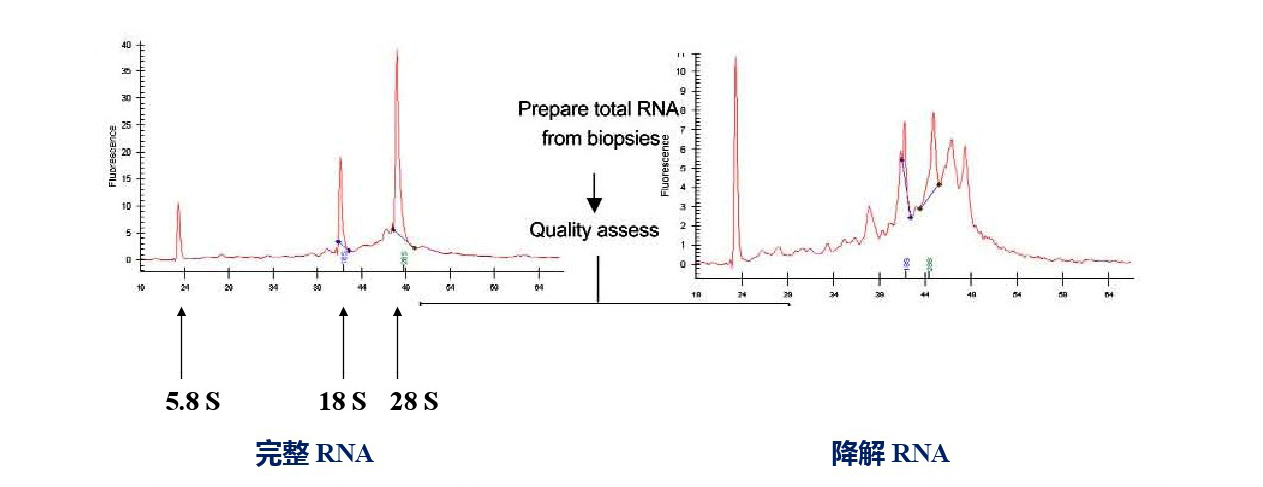
图5. 2100 or LabChip进行RNA的完整性检测。
对于降解的核酸样本来说,我们在实验中应该注意哪些点呢?特别是针对于少而珍贵的肿瘤FFPE样本或者是法医样本,这些样本中核酸的质量都比较差,降解程度较为严重,对于这些样本的qPCR实验,我们可以减少扩增产物的片段来减小样本本身给实验造成的影响。
这里也给大家列出了不同质检方法的优缺点:
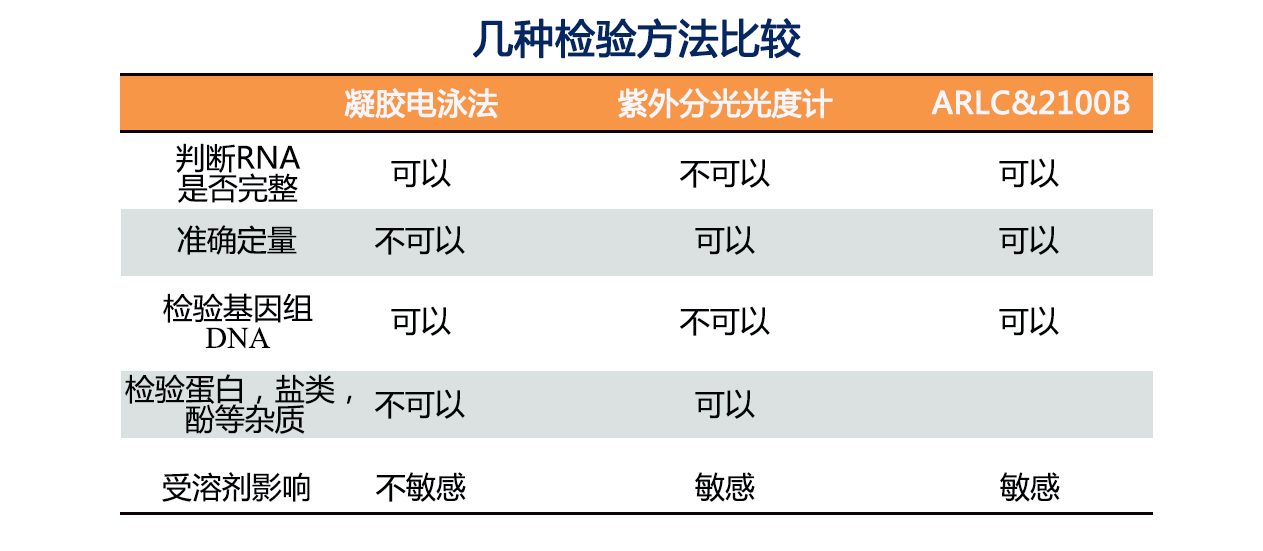
Ps:我们将继续在通过微信平台送出技术文章,包括:逆转录的解决方案,数据分析这一系列的主题;下一期的主题是逆转录的解决方案,敬请期待。